Evolusjonære hindringer for intronenes opprinnelse
 Av Jonathan McLatchie, 18. juni 2025. Oversatt herfra
Av Jonathan McLatchie, 18. juni 2025. Oversatt herfra
En stor hindring for enhver forklaring av eukaryoters opprinnelse, er fremveksten av de ikke-kodende regionene i gener kjent som introner, som i stor grad er fraværende i bakterier. Før vi diskuterer hvordan introners opprinnelse utgjør en betydelig utfordring for evolusjonen, er det først nødvendig å gi en kort introduksjon til hva introner er.
Bilde 1. Ikke-(protein)-kodende Introner skiller kodende eksoner
Hva er introner?
Introner (som, i motsetning til eksoner, ikke koder for proteiner) kan være av betydelig lengde i høyere eukaryoter, til og med strekke seg over mange tusen baser og noen ganger utgjøre rundt 90 prosent av forløperen mRNA. (1) I motsetning til dette har lavere eukaryoter som gjær, færre og kortere introner, som vanligvis er færre enn 300 baser lange. Siden introner er de ikke-kodende segmentene av gener, fjernes de fra mRNA før det oversettes til et protein. Intronene fjernes fra pre-mRNA-transkriptet, og eksonene skjøtes sammen av et riboproteinkompleks kalt spliceosomet.(2,3) Denne bemerkelsesverdige prosessen illustreres av animasjonen nedenfor:
Hvilken funksjonell rolle spiller introner?
Introner ble generelt antatt å være ikke-funksjonelt søppel-DNA da de ble oppdaget.(4,5,6,7) Francis Crick beskrev disse sekvensene som "tullete" DNA-strekninger ispedd det virkningsfulle-DNAet. (8) Dette synet på introner har imidlertid ikke tålt alderen godt. Faktisk "utfyller introner i moderne arter et bredt spekter av funksjoner og er involvert i praktisk talt alle trinn i mRNA-prosessering." (9) Introner viser ofte høye nivåer av sekvenskonservering (spesielt innenfor klasser av organismer), noe som tyder på funksjon. (10,11)
Vi vet nå også at introner spiller en essensiell rolle i alternativ spleising der eksonene i pre-mRNA-transkripter spleises i forskjellige konfigurasjoner og produserer flere proteinisoformer fra samme gen.(12,13) I følge en artikkel er majoriteten av alternativt spleisede eksoner hos mennesker og mus flankert av introner, hvis sekvenser er svært konserverte, noe som antyder en viktig rolle i alternativ spleising.(14)
Bilde 2. Spliceosomet skjøter og spleiser DNA
En annen studie dokumenterte at en 6-nukleotid intronsekvens "ofte er lokalisert ved siden av vevsspesifikke alternative eksoner i det menneskelige genomet."(15) På tvers av taxa så vidt forskjellige som hunder, rotter, kyllinger, mus og mennesker, viser denne sekvensen konservering. Disse funksjonene, ifølge artikkelen, "markerer det som en kritisk komponent i spleisingsbryter-mekanisme(r) designet for å aktivere et begrenset repertoar av spleisingshendelser i celletypespesifikke mønstre." I tillegg er det dokumentert direkte bevis som viser at introner inneholder andre koder involvert i reguleringen av alternativ spleising.(16,17,18,19,20)
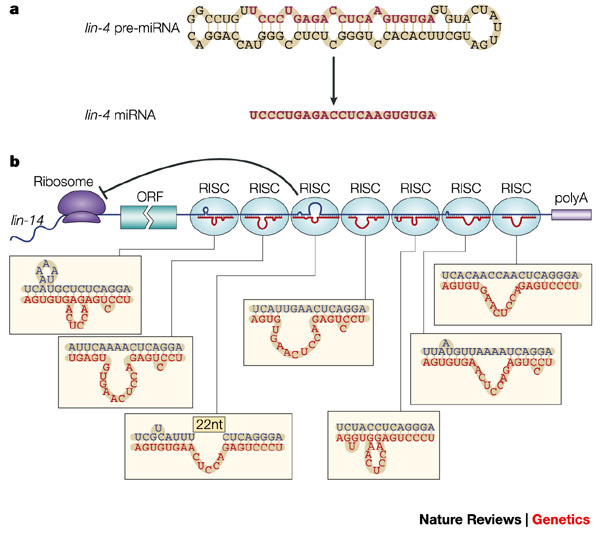 Gener for de fleste mikroRNA-er (som er nødvendige for uttrykk av mRNA-er under utvikling) og små nukleolære RNA-er (som spiller viktige roller i prosesseringen av ribosomale RNA-er) befinner seg i introner.(21) En artikkel rapporterte at:
Gener for de fleste mikroRNA-er (som er nødvendige for uttrykk av mRNA-er under utvikling) og små nukleolære RNA-er (som spiller viktige roller i prosesseringen av ribosomale RNA-er) befinner seg i introner.(21) En artikkel rapporterte at:
"Mirtroner er alternative forløpere for mikroRNA-biogenese som nylig ble beskrevet hos virvelløse dyr. Disse korte hårnålsintronene bruker spleising for å omgå Drosha-spaltning, som ellers er essensielt for generering av kanoniske dyremikroRNA-er. Ved hjelp av beregningsmessige og eksperimentelle strategier, fastslår vi nå at pattedyr også har mirtroner." (22)
Bilde 3. micro-RNA
En annen studie rapporterer også at: "314 miRNA-er (51 %) var lokalisert i introner i proteinkodende gener, 231 miRNA-er (38 %) var lokalisert i intergeniske regioner, mens bare 43 miRNA-er (7 %) var lokalisert i eksoner av ikke-kodende RNA-er eller UTR av proteinkodende gener. Interessant nok var 23 miRNA-er (4 %) lokalisert i enten et ekson eller et intron, avhengig av alternativ spleising av vertstranskriptet." (23)
Det har også blitt fastslått at promotorsekvensene for disse RNA-genene forekommer i introner.(24) Videre finnes så mange som 52 prosent av transkriptene for kromatinassosiert RNA, som spiller viktige roller i den strukturelle organiseringen av kjernekromatin (25), i introner.(26)
Det er sannsynlig at lengden på introner (og følgelig tiden det tar å transkribere dem) kan bidra til tidsmekanismer under utviklingen.(27) Siden lange introner tar lengre tid å transkribere, kan dette føre til at gener reagerer saktere på transkripsjonelle aktivatorer eller repressorer sammenlignet med kortere gener, noe som resulterer i sekvensiell genaktivering, der gener med lengre introner uttrykkes senere selv om de mottar samme aktiveringssignal samtidig.
Å forklare opprinnelsen til introner er en utfordring for evolusjonen
Hvordan kan introner oppstå i evolusjonen uten samtidig å ha en mekanisme for hånden for å legge til rette for fjerning av dem fra pre-mRNA-transkriptet? Uten tilstedeværelsen av en slik mekanisme ville intronene rote rundt de kodende områdene i genet, og selv bli oversatt til en sekvens av aminosyrer av ribosomet. Dessuten, hvis lengden på et intron ikke er et multiplum av tre, kan det også potensielt introdusere et skadelig rammeskift i den kodende regionen.
Selvspleiende introner?
Den vanligste forklaringen på opprinnelsen til eukaryote introner er at de oppsto fra selvspleiende forløpere, kjent som gruppe II-introner som finnes i bakterier.(28,29,30,31) Selvspleiende introner benytter en mekanisme som er veldig lik eukaryot pre-mRNA-spleising, som involverer to sekvensielle transesterifiseringsreaksjoner. Det første trinnet er at et konservert "forgreningspunkt"-adenosin initierer en nukleofil reaksjon ved 5'-spleisingsstedet, noe som fører til spalting av RNA-ryggraden og dannelse av en lariatstruktur. I det andre trinnet angripes 3'-spleisingsstedet av den terminale 3'-hydroksylgruppen i eksonet ved 5'-enden. Resultatet er fjerning av lariat-intronet og ligering av de flankerende eksonene.
 Det tilbys noen forskjellige bevislinjer som støtter hypotesen om at eukaryote spleiseosomale introner oppsto fra gruppe II-introner. For eksempel ville en enklere måte å oppnå spleising på antagelig være å bringe spleisingsstedene sammen i ett trinn for å direkte spalte og sammenføye dem. Det foreslåtte scenariet ville forklare bruken av et lariat-mellomprodukt, siden et lariat genereres av gruppe II RNA-intronsekvenser.(32,33) Videre argumenteres det for at denne hypotesen bidrar til å avklare hvorfor RNA-molekyler spiller en så viktig rolle i spleisingsprosessen. Eksempler på selvspleisende RNA-introner eksisterer fortsatt i dag, slik som i de nukleære rRNA-genene til ciliaten Tetrahymena.(34,35,36,37)
Det tilbys noen forskjellige bevislinjer som støtter hypotesen om at eukaryote spleiseosomale introner oppsto fra gruppe II-introner. For eksempel ville en enklere måte å oppnå spleising på antagelig være å bringe spleisingsstedene sammen i ett trinn for å direkte spalte og sammenføye dem. Det foreslåtte scenariet ville forklare bruken av et lariat-mellomprodukt, siden et lariat genereres av gruppe II RNA-intronsekvenser.(32,33) Videre argumenteres det for at denne hypotesen bidrar til å avklare hvorfor RNA-molekyler spiller en så viktig rolle i spleisingsprosessen. Eksempler på selvspleisende RNA-introner eksisterer fortsatt i dag, slik som i de nukleære rRNA-genene til ciliaten Tetrahymena.(34,35,36,37)
Det er usannsynlig at gruppe II-introner oppsto i proteinkodende gener uten eksisterende spleisemekanismer. For det første folder gruppe II-introner seg inn i en konservert sekundærstruktur med seks domener, som er nødvendig for deres selvreplikerende evne.(38) Riktig folding avhenger av spesifikk intra- og interdomenbaseparing. Dermed er nøkkelregioner ganske sekvensfølsomme. Det mest konserverte domenet er domene V, som danner det katalytiske senteret til ribozymet.(39,40) Selv enkeltnukleotidmutasjoner kan oppheve eller redusere spleiseaktiviteten betydelig. Domene I inneholder ekson-bindingssteder som må danne komplementære basepar med intronbindingssteder på eksonene. Det finnes også spesifikke sekvenser som medierer langtrekkende interaksjoner, som er essensielle for riktig folding. Gitt behovet for sekvensspesifisitet, måtte de riktige sekvensene genereres ved tilfeldige mutasjoner - men man ville forvente at introduksjonen av ytterligere sekvenser i proteinkodende gener ville være skadelig for organismen, og dermed fjernes ved rensende seleksjon.
Bilde 4. Spliceosom i aksjon
På grunn av dette antas det generelt at gruppe II-introner først må ha oppstått i ikke-kodende regioner, noe som ga en tryggere "sandkasse" for disse elementene til å utvikle den komplekse sekundærstrukturen og selvspleisingsevnen, uten umiddelbare skadelige effekter. Bakteriegenomer er imidlertid vanligvis gentette med  relativt lite ikke-kodende DNA. Dette reiser spørsmålet om hvor mye "trygt rom" som ville ha vært tilgjengelig for opprinnelsen av gruppe II-introner før de ble satt inn i kodende regioner. For eksempel har E. coli-genomet bare omtrent 10-12 % ikke-kodende DNA (~460 000-550 000 basepar DNA). Et gjennomsnittlig gruppe II-intron er ~2500 basepar langt. Dette gir bare plass til noen få dusin gruppe II-introner i full lengde å eksistere i ikke-kodende DNA uten å forstyrre verts-egnetheten.
relativt lite ikke-kodende DNA. Dette reiser spørsmålet om hvor mye "trygt rom" som ville ha vært tilgjengelig for opprinnelsen av gruppe II-introner før de ble satt inn i kodende regioner. For eksempel har E. coli-genomet bare omtrent 10-12 % ikke-kodende DNA (~460 000-550 000 basepar DNA). Et gjennomsnittlig gruppe II-intron er ~2500 basepar langt. Dette gir bare plass til noen få dusin gruppe II-introner i full lengde å eksistere i ikke-kodende DNA uten å forstyrre verts-egnetheten.
Bilde 5. Kan introner være selvspleisende?
Selv med denne hypotesen virker det imidlertid usannsynlig at det i en ikke-kodende region ville være et selektivt press for å bevare slike svært begrensede sekvensfunksjoner inntil hele intronet, komplett med sin selvspleisingsevne, var fullt ut realisert. Videre, for at spleising skal lykkes, må gruppe II-introner nøyaktig gjenkjenne ekson-intron-grenser og binde komplementære sekvenser i de flankerende eksonene. Men sandkassehypotesen vil ha oss til å tro at introner utviklet seg fullstendig innenfor ikke-kodende sekvenser, uten de nødvendige eksoniske kontekstene for å utvikle denne koordinerte interaksjonen. Selv om vi antar at et fullt operativt gruppe II-intron skulle oppstå i en ikke-kodende region, ville dets innsetting i et kodende gen være svært forstyrrende, med mindre presis spleising allerede var operativ, og nøyaktig matchet med de flankerende eksonene.
Det finnes heller ikke noe plausibelt scenario for overgang fra gruppe II-introner til spliceosomale introner. Spliceosommaskineriet er langt mer komplekst og sofistikert enn autokatalytiske ribozymer, og involverer ikke bare fem RNA-er, men nesten hundre kjerne-spliceosomproteiner som finnes i de enkleste eukaryotene.
Eukaryoter og prokaryoter deler sannsynligvis ikke en felles stamfar
 Som tidligere argumentert -lenke, viser forskjellen i celledelingsmåte mellom eukaryoter og prokaryoter deres separate opprinnelse. (41) Forekomsten av introner i eukaryote genomer, som i stor grad er fraværende i prokaryoter, gir enda en grunn til å mistenke at eukaryoter og prokaryoter hadde uavhengig opprinnelse. Nåværende forslag spekulerer i at introner i en proto-eukaryot kan ha oppstått etter infeksjon av en alfa-proteobakteriell endosymbiont (den hypotetiske mekanismen der mitokondrier og kloroplaster oppsto i eukaryoter).(42) Gruppe II intron-retroelementer som stammer fra endosymbionten, kan ha satt seg inn i det tidlige vertsgenomet.
Som tidligere argumentert -lenke, viser forskjellen i celledelingsmåte mellom eukaryoter og prokaryoter deres separate opprinnelse. (41) Forekomsten av introner i eukaryote genomer, som i stor grad er fraværende i prokaryoter, gir enda en grunn til å mistenke at eukaryoter og prokaryoter hadde uavhengig opprinnelse. Nåværende forslag spekulerer i at introner i en proto-eukaryot kan ha oppstått etter infeksjon av en alfa-proteobakteriell endosymbiont (den hypotetiske mekanismen der mitokondrier og kloroplaster oppsto i eukaryoter).(42) Gruppe II intron-retroelementer som stammer fra endosymbionten, kan ha satt seg inn i det tidlige vertsgenomet.
Bilde 6. Stor forskjell på eukaryoter (øverst) og bakterier (nederst)
Dette scenariet er svært usannsynlig, ettersom spliceosomale introner i eukaryoter viser ubetydelig likhet med gruppe II-introner og allerede ville kreve operative spliceosomer i denne tidlige evolusjonsfasen for å fjerne dem fra mRNA. Dermed er opprinnelsen til disse forskjellige intronene og mengden av spliceosomkomponenter som kreves for fjerning av dem, utilstrekkelig beskrevet av denne fremtredende evolusjonære modellen. En alternativ vurdering av intronopprinnelse beskriver syv mekanismer som ser ut til å gi opphav til sjeldne, isolerte introner, men det innrømmes at dette ikke er nok til å forklare det store flertallet av spliceosomale introner i eukaryoter.(43) Utvilsomt formidler dette sistnevnte synspunktet mer nøyaktig det vi har lært om potensielle evolusjonære forklaringer på introner.
Konklusjon
I lys av problemene som er skissert i det foregående, representerer opprinnelsen til spliceosomale introner en betydelig utfordring for en evolusjonær redegjørelse for deres opprinnelse. Selv om hypotesen om at gruppe II-introner fra bakterielle endosymbionter ga opphav til eukaryote introner er den rådende forklaringen, står den overfor en rekke betydelige hindringer. Kompleksiteten i intronspleisingsmekanismer antyder at deres opprinnelse bedre kan forklares av et annet paradigme enn ikke-styrte evolusjonære prosesser - et som tillater rask fremvekst av koordinerte, informasjonsrike systemer. Den eneste typen forklaring som tilstrekkelig forklarer denne typen fenomen er en målrettet, eller intelligent, årsak.
For Referanser, se slutten av originalartikkelen -lenke

Bilde 7. Jonathan McLatchie
RESIDENT BIOLOG & stipendiat, SENTER FOR VITENSKAP OG KULTUR
Dr. Jonathan McLatchie har en bachelorgrad i rettsmedisinsk biologi fra University of Strathclyde, en mastergrad (M.Res) i evolusjonsbiologi fra University of Glasgow, en andre mastergrad i medisinsk og molekylær biovitenskap fra Newcastle University, og en doktorgrad i evolusjonsbiologi fra Newcastle University. Tidligere var Jonathan assisterende professor i biologi ved Sattler College i Boston, Massachusetts. Jonathan har blitt intervjuet på podcaster og radioprogrammer, inkludert "Unbelieveable?" på Premier Christian Radio og mange andre. Jonathan har snakket internasjonalt i Europa, Nord-Amerika, Sør-Afrika og Asia for å fremme beviset for design i naturen.
Oversettelse, via google oversetter, og bilder, ved Asbjørn E. Lund
 Av Jonathan McLatchie, 18. juni 2025. Oversatt herfra
Av Jonathan McLatchie, 18. juni 2025. Oversatt herfra Av Jonathan McLatchie, 18. juni 2025. Oversatt herfra
Av Jonathan McLatchie, 18. juni 2025. Oversatt herfra Video: RNA-spleising
Video: RNA-spleising  Gener for de fleste mikroRNA-er (som er nødvendige for uttrykk av mRNA-er under utvikling) og små nukleolære RNA-er (som spiller viktige roller i prosesseringen av ribosomale RNA-er) befinner seg i introner.(21) En artikkel rapporterte at:
Gener for de fleste mikroRNA-er (som er nødvendige for uttrykk av mRNA-er under utvikling) og små nukleolære RNA-er (som spiller viktige roller i prosesseringen av ribosomale RNA-er) befinner seg i introner.(21) En artikkel rapporterte at: Det tilbys noen forskjellige bevislinjer som støtter hypotesen om at eukaryote spleiseosomale introner oppsto fra gruppe II-introner. For eksempel ville en enklere måte å oppnå spleising på antagelig være å bringe spleisingsstedene sammen i ett trinn for å direkte spalte og sammenføye dem. Det foreslåtte scenariet ville forklare bruken av et lariat-mellomprodukt, siden et lariat genereres av gruppe II RNA-intronsekvenser.(32,33) Videre argumenteres det for at denne hypotesen bidrar til å avklare hvorfor RNA-molekyler spiller en så viktig rolle i spleisingsprosessen. Eksempler på selvspleisende RNA-introner eksisterer fortsatt i dag, slik som i de nukleære rRNA-genene til ciliaten Tetrahymena.(34,35,36,37)
Det tilbys noen forskjellige bevislinjer som støtter hypotesen om at eukaryote spleiseosomale introner oppsto fra gruppe II-introner. For eksempel ville en enklere måte å oppnå spleising på antagelig være å bringe spleisingsstedene sammen i ett trinn for å direkte spalte og sammenføye dem. Det foreslåtte scenariet ville forklare bruken av et lariat-mellomprodukt, siden et lariat genereres av gruppe II RNA-intronsekvenser.(32,33) Videre argumenteres det for at denne hypotesen bidrar til å avklare hvorfor RNA-molekyler spiller en så viktig rolle i spleisingsprosessen. Eksempler på selvspleisende RNA-introner eksisterer fortsatt i dag, slik som i de nukleære rRNA-genene til ciliaten Tetrahymena.(34,35,36,37) relativt lite ikke-kodende DNA. Dette reiser spørsmålet om hvor mye "trygt rom" som ville ha vært tilgjengelig for opprinnelsen av gruppe II-introner før de ble satt inn i kodende regioner. For eksempel har E. coli-genomet bare omtrent 10-12 % ikke-kodende DNA (~460 000-550 000 basepar DNA). Et gjennomsnittlig gruppe II-intron er ~2500 basepar langt. Dette gir bare plass til noen få dusin gruppe II-introner i full lengde å eksistere i ikke-kodende DNA uten å forstyrre verts-egnetheten.
relativt lite ikke-kodende DNA. Dette reiser spørsmålet om hvor mye "trygt rom" som ville ha vært tilgjengelig for opprinnelsen av gruppe II-introner før de ble satt inn i kodende regioner. For eksempel har E. coli-genomet bare omtrent 10-12 % ikke-kodende DNA (~460 000-550 000 basepar DNA). Et gjennomsnittlig gruppe II-intron er ~2500 basepar langt. Dette gir bare plass til noen få dusin gruppe II-introner i full lengde å eksistere i ikke-kodende DNA uten å forstyrre verts-egnetheten. Som tidligere argumentert -lenke, viser forskjellen i celledelingsmåte mellom eukaryoter og prokaryoter deres separate opprinnelse. (41) Forekomsten av introner i eukaryote genomer, som i stor grad er fraværende i prokaryoter, gir enda en grunn til å mistenke at eukaryoter og prokaryoter hadde uavhengig opprinnelse. Nåværende forslag spekulerer i at introner i en proto-eukaryot kan ha oppstått etter infeksjon av en alfa-proteobakteriell endosymbiont (den hypotetiske mekanismen der mitokondrier og kloroplaster oppsto i eukaryoter).(42) Gruppe II intron-retroelementer som stammer fra endosymbionten, kan ha satt seg inn i det tidlige vertsgenomet.
Som tidligere argumentert -lenke, viser forskjellen i celledelingsmåte mellom eukaryoter og prokaryoter deres separate opprinnelse. (41) Forekomsten av introner i eukaryote genomer, som i stor grad er fraværende i prokaryoter, gir enda en grunn til å mistenke at eukaryoter og prokaryoter hadde uavhengig opprinnelse. Nåværende forslag spekulerer i at introner i en proto-eukaryot kan ha oppstått etter infeksjon av en alfa-proteobakteriell endosymbiont (den hypotetiske mekanismen der mitokondrier og kloroplaster oppsto i eukaryoter).(42) Gruppe II intron-retroelementer som stammer fra endosymbionten, kan ha satt seg inn i det tidlige vertsgenomet. 